In deze gids zullen we eerst de aanpak zien en vervolgens de beroerte van elke persoon voorspellen door de gegevens stap voor stap te analyseren.
Logistieke regressie
Logistische regressie, ook wel het 'Logit-model' genoemd, wordt gebruikt in voorspellende analyses voor classificatieproblemen. Dit is een Machine Learning-model dat alleen werkt op classificatie om de klassen (categorieën) te classificeren. Er zijn bijvoorbeeld twee categorieën in ons scenario (een persoon die getroffen is door een beroerte en een persoon die geen beroerte heeft). De beste toepassingen van het model zijn voorspelling van hartziekten, geslachtsvoorspelling, voorspelling van de levensduur van gewassen, enz.
Stappen:
1. Gegevensverzameling: De gegevens zijn nodig om eventuele voorspellingen/analyses uit te voeren. Dit kan onder andere de vorm van CSV/XLSX hebben. We kunnen dit in de Spark-omgeving (DataFrame) laden met behulp van de methode spark.read.csv().
2. Gegevensanalyse : Het analyseren van de attributen/kolommen staat bekend als “data-analyse”. De kolommen die helpen om de klasse te voorspellen, staan bekend als 'onafhankelijke attributen'. De kolom die resulteert in voorspelling staat bekend als 'afhankelijk of doelattribuut'. In dit scenario kunnen we de eigenschap columns gebruiken om alle kolommen weer te geven. De distinct() methode wordt gebruikt om de unieke waarden te zien.
3. Gegevensvoorverwerking: Het filteren van de null/ontbrekende waarden staat bekend als 'preprocessing'. We verwijderen alle ontbrekende waarden in deze fase. De machine kent alleen de binaire taal. Alle tekenreekscategorieën moeten dus worden geconverteerd naar numerieke categorische waarden. In PySpark kunnen we de 'StringIndexer', de beschikbare klasse in de module pyspark.ml.feature, gebruiken om de tekenreekscategorieën naar numeriek te converteren. Het converteert ze automatisch intern. We hoeven de waarden niet op te geven. U kunt de volgende syntaxis gebruiken:
indexer_data=StringIndexer(inputCol= 'String_Category_ColumnName' ,outputCol= 'Nieuwe_kolomnaam' )
4. Vectormontage: Nu hebt u de gegevens die de machine kan begrijpen. In deze fase moeten alle onafhankelijke attributen in een enkele kolom worden gevectoriseerd. Dit kan worden gedaan met behulp van de klasse VectorAssembler. Er zijn twee parameters voor nodig: de eerste parameter is de inputCols die een lijst met onafhankelijke attributen nodig heeft. De tweede parameter is de outputCol die alle inputCols in deze variabele vectoriseert.
assembler=VectorAssembler(inputCols=[kolommen...],outputCol=vectorized_data)5. Transformatie: Bereid nu uw gegevens voor door de bijgewerkte kolommen te transformeren (stap 3) met behulp van de functie transform().
assembler.transform(inxed_data)
6. De gegevens voorbereiden voor training en testen: In deze fase splitsen we de data op in “training” en “testen”. Het is beter als we de 70% data splitsen om het model te trainen en 30% data om het model te testen. Dit kan worden bereikt met de methode randomSplit(). Er is een lijst voor nodig die twee float-waarden bevat: een voor testsplitsing en een andere voor treinsplitsing.
train_data,test_data=final_data.select([ 'functies' ,target_column]).randomSplit([0.70.0.30])7. Modelaanpassing & Evaluatie : Het is tijd om het Logistic Regression-model te passen. Het Logistic Regression-model is beschikbaar in de module pyspark.ml.classification. Hiervoor is de klasselabel/doelkolom nodig. Dit resulteert in rawPrediction-, waarschijnlijkheids- en voorspellingskolommen. Onze resultaten worden opgeslagen in de voorspellingskolom.
# Modelpassinglogistics_regression_model=LogisticRegression(labelCol=target_column).fit(train_data)
# Modelevaluatie
train_results=logistic_regression_model.evaluate(train_data).predictions
8. Nauwkeurigheid en resultaten: Dit is de laatste fase waarin we de modelnauwkeurigheid testen met behulp van testtechnieken.
Laten we eens kijken naar het volgende project waarin we voorspellen of een persoon door een beroerte wordt getroffen of niet met behulp van het Logistic Regression-model.
Beroerte voorspelling
9. Installeer de PySpark-module in uw omgeving. Het volgende is de opdracht om te installeren:
pip installeer pyspark 
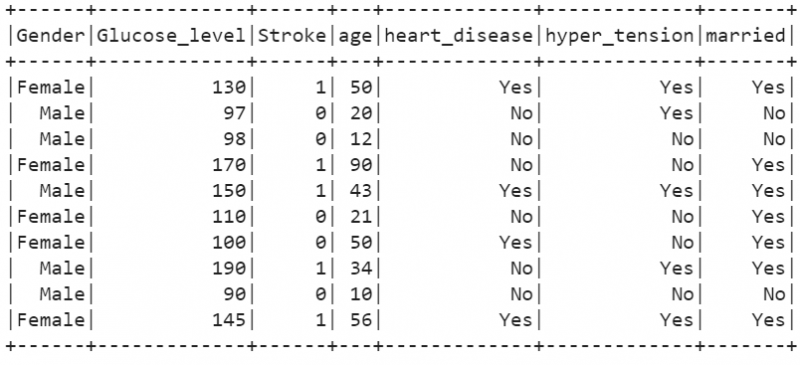
10. Laten we een PySpark DataFrame maken met 10 records die betrekking hebben op beroertegegevens van 10 personen. Ter demonstratie maken we een DataFrame zonder CSV. Er zijn 7 kolommen in dit DataFrame. 'Geslacht', 'Glucose_niveau', 'getrouwd', 'leeftijd', 'hartziekte', 'hyper_tensie' zijn onafhankelijke attributen en 'Beroerte' is het klassenlabel of afhankelijke attribuut. Dat betekent dat de slag van een persoon afhankelijk is van deze onafhankelijke attributen.
pyspark importerenimporteer vanuit pyspark.sql SparkSession
linuxhint_spark_app = SparkSession.builder.appName( 'Linux-tip' ).getOrCreate()
stoke_data =[
{ 'Geslacht' : 'Vrouwelijk' , 'leeftijd' : vijftig , 'hartziekte' : 'Ja' ,
'hyper_tensie' : 'Ja' , 'getrouwd' : 'Ja' , 'Glucose_niveau' : 130 , 'Hartinfarct' : 1 },
{ 'Geslacht' : 'Mannelijk' , 'leeftijd' : twintig , 'hartziekte' : 'Nee' ,
'hyper_tensie' : 'Ja' , 'getrouwd' : 'Nee' , 'Glucose_niveau' : 97 , 'Hartinfarct' : 0 },
{ 'Geslacht' : 'Mannelijk' , 'leeftijd' : 12 , 'hartziekte' : 'Nee' ,
'hyper_tensie' : 'Nee' , 'getrouwd' : 'Nee' , 'Glucose_niveau' : 98 , 'Hartinfarct' : 0 },
{ 'Geslacht' : 'Vrouwelijk' , 'leeftijd' : 90 , 'hartziekte' : 'Nee' ,
'hyper_tensie' : 'Nee' , 'getrouwd' : 'Ja' , 'Glucose_niveau' : 170 , 'Hartinfarct' : 1 },
{ 'Geslacht' : 'Mannelijk' , 'leeftijd' : 43 , 'hartziekte' : 'Ja' ,
'hyper_tensie' : 'Ja' , 'getrouwd' : 'Ja' , 'Glucose_niveau' : 150 , 'Hartinfarct' : 1 },
{ 'Geslacht' : 'Vrouwelijk' , 'leeftijd' : eenentwintig , 'hartziekte' : 'Nee' ,
'hyper_tensie' : 'Nee' , 'getrouwd' : 'Ja' , 'Glucose_niveau' : 110 , 'Hartinfarct' : 0 },
{ 'Geslacht' : 'Vrouwelijk' , 'leeftijd' : vijftig , 'hartziekte' : 'Ja' ,
'hyper_tensie' : 'Nee' , 'getrouwd' : 'Ja' , 'Glucose_niveau' : 100 , 'Hartinfarct' : 0 },
{ 'Geslacht' : 'Mannelijk' , 'leeftijd' : 3. 4 , 'hartziekte' : 'Nee' ,
'hyper_tensie' : 'Ja' , 'getrouwd' : 'Ja' , 'Glucose_niveau' : 190 , 'Hartinfarct' : 1 },
{ 'Geslacht' : 'Mannelijk' , 'leeftijd' : 10 , 'hartziekte' : 'Nee' ,
'hyper_tensie' : 'Nee' , 'getrouwd' : 'Nee' , 'Glucose_niveau' : 90 , 'Hartinfarct' : 0 },
{ 'Geslacht' : 'Vrouwelijk' , 'leeftijd' : 56 , 'hartziekte' : 'Ja' ,
'hyper_tensie' : 'Ja' , 'getrouwd' : 'Ja' , 'Glucose_niveau' : 145 , 'Hartinfarct' : 1 }
]
# maak het dataframe van de bovenstaande gegevens
stroke_df = linuxhint_spark_app.createDataFrame(stoke_data)
# Actuele stoke_df
stroke_df.show()
Uitgang:
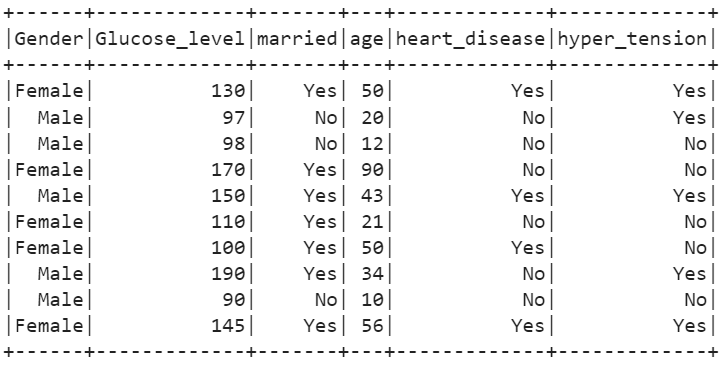
11. Geef de onafhankelijke kolommen weer met behulp van de select()-methode.
# Geef onafhankelijke attributen weerstroke_df.select(stroke_df[ 'Geslacht' ],slag_df[ 'Glucose_niveau' ],slag_df[ 'getrouwd' ],slag_df[ 'leeftijd' ],slag_df[ 'hartziekte' ],slag_df[ 'hyper_tensie' ]).show()
Uitgang:
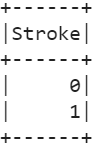
12. Geef de unieke waarden weer die aanwezig zijn in het doelattribuut (Stroke).
# doelattribuut unieke waardenstroke_df.select(stroke_df[ 'Hartinfarct' ]).distinct().show()
Uitgang:
13. Retourneer het gegevenstype van alle kolommen met behulp van de functie printSchema().
# Retourneer datatype van alle kolommen.stroke_df.printSchema()
Uitgang:

We kunnen zien dat 4 kolommen van het type string zijn. Laten we ze omzetten in categorische numerieke waarden.
14. Laten we de categorische tekenreekswaarden converteren naar numerieke categorische waarden met behulp van de StringIndexer in de kolommen 'Gender', 'heart_disease', 'hyper_tension' en 'married' en deze in de kolommen Categotical_Gender, Categotical_heart_disease, Categotical_hyper_tension en Categotical_married schrijven. Sla de kolommen op in het geïndexeerde_data DataFrame met behulp van de fit() methode.
van pyspark.ml.feature importeer StringIndexer# Converteer categorische tekenreekswaarden naar numerieke categorische waarden in de kolom 'Geslacht'.
indexer_data=StringIndexer(inputCol= 'Geslacht' ,outputCol= 'Categotisch_geslacht' )
geïndexeerde_data=indexer_data.fit(stroke_df).transform(stroke_df)
# Converteer string categorische waarden naar numerieke categorische waarden in de kolom 'heart_disease'.
indexer_data=StringIndexer(inputCol= 'hartziekte' ,outputCol= 'Categotische_hartziekte' )
geïndexeerde_data=indexer_data.fit(geïndexeerde_data).transform(geïndexeerde_data)
# Converteer string categorische waarden naar numerieke categorische waarden in de kolom 'hyper_tension'.
indexer_data=StringIndexer(inputCol= 'hyper_tensie' ,outputCol= 'Categotische_hyper_spanning' )
geïndexeerde_data=indexer_data.fit(geïndexeerde_data).transform(geïndexeerde_data)
# Converteer categorische tekenreekswaarden naar numerieke categorische waarden in de kolom 'getrouwd'.
indexer_data=StringIndexer(inputCol= 'getrouwd' ,outputCol= 'Categotisch_getrouwd' )
geïndexeerde_data=indexer_data.fit(geïndexeerde_data).transform(geïndexeerde_data)
# Geef de bijgewerkte weer
geïndexeerde_data.show()
Uitgang:
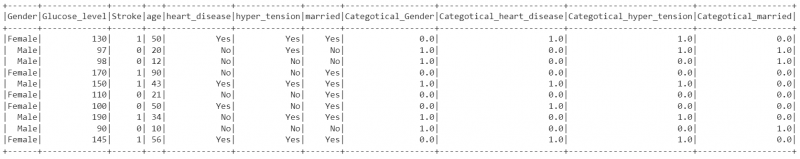
15. Converteer de onafhankelijke kolommen naar een vector met behulp van de vector-assembler. De naam van de vector is 'kenmerken'.
van pyspark.ml.feature importeer VectorAssemblerassembler= VectorAssembler(inputCols=[ 'Categotisch_geslacht' , 'Categotische_hartziekte' , 'Categotische_hyper_spanning' , 'Categotisch_getrouwd' , 'leeftijd' ,
'Glucose_niveau' ],outputCol= 'functies' )
16. Transformeer de vorige gegevens in een definitief DataFrame met behulp van de functie transform() en geef deze weer met de functie show().
Uitgang:

17. Bereid de gegevens voor op training en testen door deze op te splitsen in 70-30. De 'features' wordt opgeslagen in de train_data en 'Stroke' wordt opgeslagen in de test_data.
# Gegevens voorbereiden voor training en testentrain_data,test_data=final.select([ 'functies' , 'Hartinfarct' ]).randomSplit([ 0,70 , 0.30 ])
18. Pas het Logistic Regression-model aan en evalueer het.
van pyspark.ml.classification import LogisticRegression# Modelpassing
logistics_regression_model=LogisticRegression(labelCol= 'Hartinfarct' .fit(treingegevens)
# Modelevaluatie
train_results=logistic_regression_model.evaluate(train_data).predictions
train_results.show()
Uitgang:
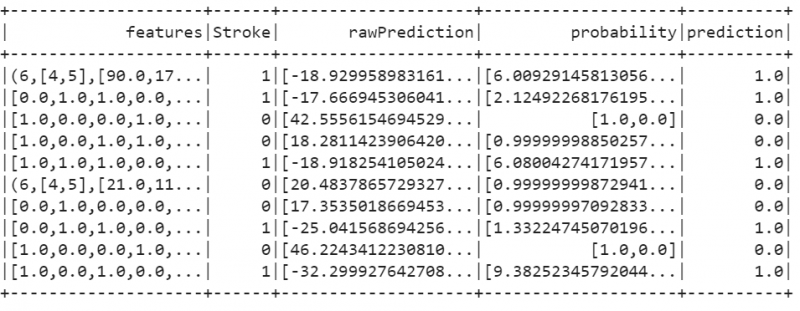
19. Gebruik de BinaryClassificationEvaluator om de modelnauwkeurigheid te controleren. We kunnen zien dat ons model 100% nauwkeurig is.
van pyspark.ml.evaluation import BinaryClassificationEvaluator# bel de BinaryClassificationEvaluator
resultaten = BinaryClassificationEvaluator(rawPredictionCol= 'voorspelling' ,labelKol= 'Hartinfarct' )
ROC_AUC = resultaten.evalueren(train_results)
afdrukken(ROC_AUC * 100 , '% Nauwkeurigheid' )
Conclusie
U kunt het real-tile classificatieprobleem voorspellen met behulp van het Logistic Regression-model in PySpark. Het verwijderen van de null/ontbrekende waarden is belangrijk omdat ze de modelevaluatie en nauwkeurigheid verminderen. Voorverwerking is een noodzakelijke stap voordat een machine learning-model wordt aangepast. Zorg ervoor dat u deze stap correct en nauwkeurig uitvoert.